【提示】本文写作命令为:abysw blog APA分析TAPIS
总的来说,注释大概分如下这些部分,这只是个糙版,很久之后可能会更新。
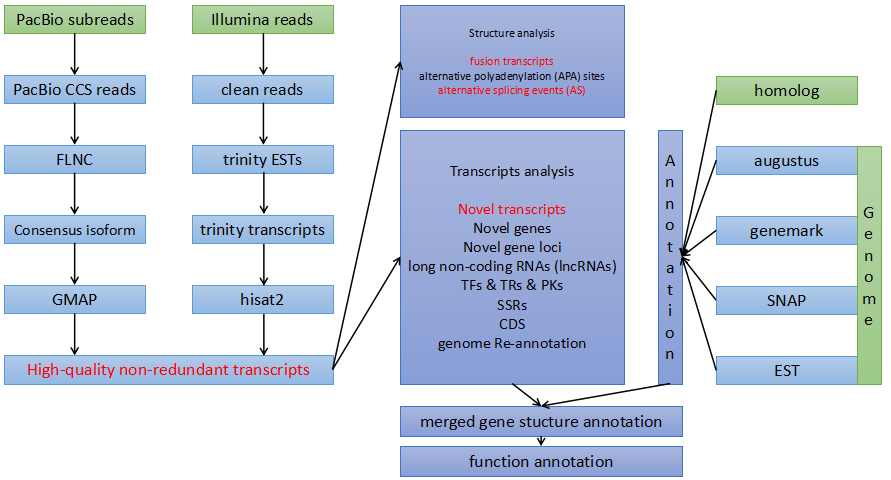
有的过程很简单,就没有写笔记了。
========================================================
APA分析的工具TAPIS,需要注意修改部分脚本,主要注意事项如下:
- 所依赖的python版本为2.7;
- fasta用非压缩文件时,需要修改代码;
- 所使用的samtools是老版本,我的版本是1.7.
========================================================
用python 2.7安装本软件,在做注释的时候注意时常切换python版本;
主脚本第79行,修改为:prefix = ‘cat ‘;
副脚本第44行修改为:
cmd = “samtools sort -@ %d -m %dG -O BAM -o %s.bam %s” %(processors, memory, sorted_basename, bamfile)
主脚本第125行:
【不用修改,两个版本的用法没变】
========================================================
alignPacBio.py -p 64 -o ./$R $PWD/ ${S}_genome ${S}_genome.fa $PWD/RFSCS.collapsed.filtered.rep.fa
我一般比对使用的都是64线程(服务器线程是256)
run_tapis.py -o $R $S.gtf $R/aligned.bam
里面还有AS和noval gene的分析结果,可以和其他软件的做以下比较。
注:近期更新的日志多为调试版本(意思就是都是踩坑版,而且不一定趟到了所有坑)。